연세소식
-
[연구 프론티어] 송민 교수팀, 생물의학 문헌에서 개체 간 관계 유형 분류 모델 개발
연세대학교 홍보팀 / news@yonsei.ac.kr2022-05-24 -
송민 교수팀, 생물의학 문헌에서 개체 간 관계 유형 분류 모델 개발
대규모 학습 데이터셋 구축 및 딥러닝 기반 알고리즘 성능 심층 평가

[사진 1. 송민 교수]
문과대학 문헌정보학과 송민 교수 연구팀은 생물의학 문헌에서 바이오 개체 간 관계를 특정 서술어 유형 중 하나로 분류해 내는 딥러닝 기반 관계 유형 분류 모델을 개발했다.
최근 생물의학 분야의 연구 성과는 기하급수적으로 증가하고 있다. 구조화되지 않은 대량의 데이터에서 중요 정보를 정제하는 과정에서는 관계 추출이 핵심적인 역할을 수행한다. 공중보건 증진을 위해 약물 간 상호작용(drug-drug interaction, DDI), 단백질 간 상호작용(protein-protein interaction, PPI), 화합물-단백질 관계(chemical compound-protein relation, CPR), 유전자-질병 관계(gene-disease relation) 등을 규명하려는 연구가 활발하게 진행되고 있다.
기존 생물의학 정보추출 연구에서는 주로 규칙 기반 모델이나 기계학습 기반 접근 방식을 활용했는데, 이러한 방법은 특질(feature) 처리 측면에서 효율적이지 않고 정확도가 낮을 수 있다. 기존의 관계 추출 시스템은 신경망 기반 CNN이나 RNN 등을 활용했지만, 관계 유형 간 성능이 차이 나는 이유를 명확히 분석하지 않았다. 또한, 관계 분류에 활용할 수 있는 구조화된 데이터셋이 충분하지 않았다.
이와 같은 문제를 해결하기 위해 연구팀은 대규모 학습 데이터셋을 구축하고 딥러닝 기반 알고리즘 성능을 평가해 생물의학 관계 유형 분류에 최적화된 모델을 확인했다. 추가로 FrameNet 기반으로 서술어를 의미 기반 군집화해(clustering) 모델의 성능을 심층적으로 분석했다.
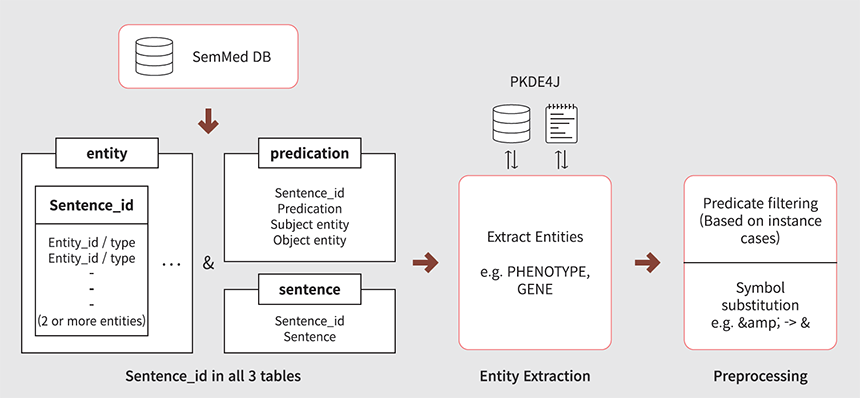
[사진 2. 데이터 구축 절차]
본 연구팀은 PubMed 문헌 데이터에서 추출된 주어-술어-목적어 관계의 의미적 서술부(semantic predications)의 저장소인 SemMed DB를 학습 데이터셋의 토대로 활용했다. 연구팀에서 기구축한 사전 기반 바이오 개체 추출 시스템인 PKDE4J를 활용해 사용자가 정의한 개체와 관계 유형을 추가할 수 있도록 했으며, 이렇게 구축된 최종 데이터셋은 온라인에 공개해 필요한 모든 연구자가 활용할 수 있도록 했다(https://github.com/deeptextlab/BioPREP).
성능 평가 결과, 신경망 기반 CNN이나 LSTM 모델에 비해 트랜스포머 기반 BERT 언어모델의 성능이 높은 것을 확인했고, 생물의학 말뭉치로 사전 학습시킨 BioBERT가 생물의학 관계 유형 분류에 가장 적합한 것으로 관측됐다. 또한, 군집화된 7개 그룹에 대해 성능 평가를 수행해 다른 서술부와 구별 기준이 모호하기 때문에 상태-증상 관계(Conditions Symptom Relation) 유형의 분류 성능이 상대적으로 낮은 것을 확인했다.
본 연구는 과학기술정보통신부와 한국연구재단이 추진하는 바이오·의료기술개발사업과 재단법인 전통천연물기반 유전자동의보감 사업단의 지원을 받아 수행됐다. 연구 결과는 생물의학 관련 정보학 분야의 국제저명학술지 ‘Journal of Biomedical Informatics(IF 6.98)’ 2021년 10월호에 온라인 게재됐다.
논문정보
● 논문제목: BioPREP: Deep learning-based predicate classification with SemMedDB
- vol. 전체
- vol. 635
- vol. 634
- vol. 633
- vol. 632
- vol. 631
- vol. 630
- vol. 629
- vol. 628
- vol. 627
- vol. 626
- vol. 625
- vol. 624
- vol. 623
- vol. 622
- vol. 621
- vol. 620
- vol. 619
- vol. 618
- vol. 617
- vol. 616
- vol. 615
- vol. 614
- vol. 613
- vol. 612
- vol. 611
- vol. 610
- vol. 609
- vol. 608
- vol. 607
- vol. 606
- vol. 605
- vol. 604
- vol. 603
- vol. 602
- vol. 601
- vol. 600
- vol. 599
- vol. 598
- vol. 597
- vol. 596
- vol. 595
- vol. 594
- vol. 593
- vol. 592
- vol. 591
- vol. 590
- vol. 589
- vol. 587
- vol. 586
- vol. 585
- vol. 584
- vol. 583
- vol. 582
- vol. 581
- vol. 580
- vol. 579
- vol. 578
- vol. 577
- vol. 576
- vol. 575
- vol. 574
- vol. 573
- vol. 572
- vol. 571
- vol. 570
- vol. 569
- vol. 568
- vol. 567
- vol. 566
- vol. 565
- vol. 564
- vol. 563
- vol. 562
- vol. 561
- vol. 560
- vol. 559
- vol. 558
- vol. 557
- vol. 556
- vol. 555
- vol. 554
- vol. 553
- vol. 552
- vol. 551
- vol. 550
- vol. 549
- vol. 548
- vol. 547
- vol. 546
- vol. 545
- vol. 544
- vol. 543
- vol. 542
- vol. 541
- vol. 540
- vol. 539
- vol. 538
- vol. 537
- vol. 536
- vol. 535
- vol. 534
- vol. 533
- vol. 532
- vol. 531
- vol. 530
- vol. 529
- vol. 528
- vol. 527
- vol. 526
- vol. 525
- vol. 524
- vol. 523
- vol. 522
- vol. 521
- vol. 520
- vol. 519
- vol. 518
- vol. 517
- vol. 516
- vol. 515
- vol. 514
- vol. 513
- vol. 512
- vol. 511
- vol. 510
- vol. 509
- vol. 508
- vol. 507
- vol. 506
- vol. 505
- vol. 504
- vol. 503
- vol. 502
- vol. 501
- vol. 500
- vol. 499
- vol. 498
- vol. 497
- vol. 496
- vol. 495
- vol. 494
- vol. 493
- vol. 492
- vol. 491
- vol. 490
- vol. 489
- vol. 488
- vol. 487
- vol. 486
- vol. 485
- vol. 484
- vol. 483
- vol. 482
- vol. 481
- vol. 480
- vol. 479
- vol. 478
- vol. 477
- vol. 476
- vol. 475
- vol. 474
- vol. 473
- vol. 472
- vol. 471
- vol. 470
- vol. 469
- vol. 468
- vol. 467
- vol. 466
- vol. 465
- vol. 464
- vol. 463
- vol. 462
- vol. 461
- vol. 460
- vol. 459
- vol. 458
- vol. 457
- vol. 456
- vol. 455
- vol. 454
- vol. 453
- vol. 452
- vol. 451
- vol. 450
- vol. 449
- vol. 448
- vol. 447
- vol. 446
- vol. 445
- vol. 444
- vol. 443
- vol. 442
- vol. 441
- vol. 440
- vol. 439
- vol. 438
- vol. 437
- vol. 436
- vol. 435
- vol. 434
- vol. 433
- vol. 432
- vol. 431
- vol. 430
- vol. 429
- vol. 428
- vol. 427
- vol. 426
- vol. 425
- vol. 424
- vol. 423
- vol. 422
- vol. 421
- vol. 420
- vol. 419
- vol. 418
- vol. 417
- vol. 416
- vol. 415
- vol. 414
- vol. 413
- vol. 412
- vol. 411
- vol. 410
- vol. 409
- vol. 408
- vol. 407
- vol. 406
- vol. 405
- vol. 404
- vol. 403
- vol. 402
- vol. 401
- vol. 400
- vol. 399
- vol. 398
- vol. 397
- vol. 396
- vol. 395
- vol. 394
- vol. 393
- vol. 392
- vol. 391
- vol. 390
- vol. 389
- vol. 388
- vol. 387
- vol. 386
- vol. 385
- vol. 384
- vol. 383
- vol. 382
- vol. 381
- vol. 380
- vol. 379
- vol. 378
- vol. 377
- vol. 376
- vol. 375
- vol. 374
- vol. 373
- vol. 372
- vol. 371
- vol. 370
- vol. 369
- vol. 368
- vol. 367
- vol. 366
- vol. 365
- vol. 364
- vol. 363
- vol. 362
- vol. 361
- vol. 360
- vol. 359
- vol. 358
- vol. 357
- vol. 356
- vol. 355
- vol. 354
- vol. 353
- vol. 352
- vol. 351
- vol. 350
- vol. 349
- vol. 348
- vol. 347
- vol. 346
- vol. 345
- vol. 344
- vol. 343
- vol. 342
- vol. 341
- vol. 340
- vol. 339
- vol. 338
- vol. 337
- vol. 336
- vol. 335
- vol. 334
- vol. 333
- vol. 332
- vol. 331
- vol. 330
- vol. 329
- vol. 328
- vol. 327
- vol. 326
- vol. 325
- vol. 324
- vol. 323
- vol. 322
- vol. 321
- vol. 320
- vol. 319
- vol. 318
- vol. 317
- vol. 316
- vol. 315
- vol. 314
- vol. 313
- vol. 312
- vol. 311
- vol. 310
- vol. 309
- vol. 308
- vol. 307
- vol. 306
- vol. 305
- vol. 304
- vol. 303
- vol. 302
- vol. 301
- vol. 300
- vol. 299
- vol. 298
- vol. 297
- vol. 296
- vol. 295
- vol. 294
- vol. 293
- vol. 292
- vol. 291
- vol. 290
- vol. 289
- vol. 288
- vol. 287
- vol. 286
- vol. 285
- vol. 284
- vol. 283
- vol. 282
- vol. 281
- vol. 280
- vol. 279
- vol. 278
- vol. 277
- vol. 276
- vol. 275
- vol. 274
- vol. 273
- vol. 272
- vol. 271
- vol. 270
- vol. 269
- vol. 268
- vol. 267
- vol. 266
- vol. 265
- vol. 264
- vol. 263
- vol. 262
- vol. 261
- vol. 260
- vol. 259
- vol. 258
- vol. 257
- vol. 256
- vol. 255
- vol. 254
- vol. 253
- vol. 252
- vol. 251
- vol. 250
- vol. 249
- vol. 248
- vol. 247
- vol. 246
- vol. 245
- vol. 244
- vol. 243
- vol. 242
- vol. 241
- vol. 240
- vol. 239
- vol. 238
- vol. 237
- vol. 236
- vol. 235
- vol. 234
- vol. 233
- vol. 232
- vol. 231
- vol. 230
- vol. 229
- vol. 228
- vol. 227
- vol. 226
- vol. 225
- vol. 224
- vol. 223
- vol. 222
- vol. 221
- vol. 220
- vol. 219
- vol. 218
- vol. 217
- vol. 216
- vol. 215
- vol. 214
- vol. 213
- vol. 212
- vol. 211
- vol. 210
- vol. 209
- vol. 208
- vol. 207
- vol. 206
- vol. 205
- vol. 204
- vol. 203
- vol. 202
- vol. 201
- vol. 189
- vol. 188
- vol. 187
- vol. 186
- vol. 185
- vol. 184
- vol. 183
- vol. 182
- vol. 181
- vol. 180
- vol. 179
- vol. 178
- vol. 177
- vol. 176
- vol. 175
- vol. 174
- vol. 173
- vol. 172
- vol. 171
- vol. 170
- vol. 169
- vol. 168
- vol. 167
- vol. 166
- vol. 165
- vol. 164
- vol. 163
- vol. 162
- vol. 161
- vol. 160
- vol. 159
- vol. 158
- vol. 157
- vol. 156
- vol. 155
- vol. 154
- vol. 153
- vol. 152
- vol. 151
- 전체 글 보기
- 연세 뉴스
- 여기 연세인
- 연세 사랑
- 창업톡톡
- 화제의 인물
- Academia
- 연구 프론티어
- LearnUs 이달의 강의
- 신촌캠퍼스 소식
- 의료원 소식
- 미래캠퍼스 소식
- 국제캠퍼스 소식
- 신간 안내
- 동정
연세소식 신청방법
아래 신청서를 작성 후 news@yonsei.ac.kr로 보내주세요신청서 다운로드