연세소식
-
[연구 프론티어] 이인석 교수 연구팀, 유전체코호트 네트워크 분석을 통한 질병유전자 예측 시스템 개발
연세대학교 홍보팀 / news@yonsei.ac.kr2017-06-01 -
이인석 교수 연구팀, 유전체코호트 네트워크 분석을 통한 질병유전자 예측 시스템 개발
시스템생물학 이용해 유전체코호트 빅데이터 기반 질병유전자 예측력 획기적으로 증대

이인석 교수(생명공학과) 연구팀이 최근 질병유전자 발굴에 가장 많이 사용되고 있는 대규모 유전체코호트 빅데이터를 네트워크 정보와 통합하여 고효율저비용으로 신규 질병유전자들을 발굴할 수 있는 개방형 예측 시스템 ‘GWAB’을 개발했다.
인간 게놈프로젝트가 완성된 이후 정상인과 환자의 대규모집단(cohort)을 구축하고 이들의 유전체정보를 분석·비교하여 질병에 연관된 유전체 변이를 발굴하고, 이를 기반으로 질병유전자까지도 발굴할 수 있는 연구인 GWAS(Genome-wide association study)가 다양한 질병의 연구를 위해 진행되어 왔다. 이러한 유전체 연구법은 이제까지 많은 질병유전자를 발굴하는 데 크게 기여했지만 일반적으로 수십억 원의 연구비용으로 수십 개 이하의 질병유전자만을 발굴할 수 있는 효율성의 한계가 있었다. 이러한 문제를 해결하기 위해 더 많은 사람들의 유전체를 분석하거나 여러 집단으로부터 나온 데이터를 병합하여 통계적인 민감도를 올리는 방법들이 사용되어 왔으나, 고가의 추가 연구비용이 요구되어 많은 연구자들이 쉽게 시도할 수 없는 한계가 있었다.
[그림1] 유전체코호트 빅데이터와 유전자네트워크 통합 시스템의 개념 요약도
최근 이인석 교수와 심정은 연구교수는 기존의 통계적인 문제 해결 접근법 대신에 시스템 생물학을 이용한 방법을 제시하고 이를 이용 하여 대사질환, 심혈관질환, 뇌신경질환, 면역질환 등의 다양한 질병들에 관련된 신규 유전자들을 예측함으로써 유전체코호트 빅데이터의 가치를 획기적으로 증대할 수 있는 기술적인 발판을 마련했다.

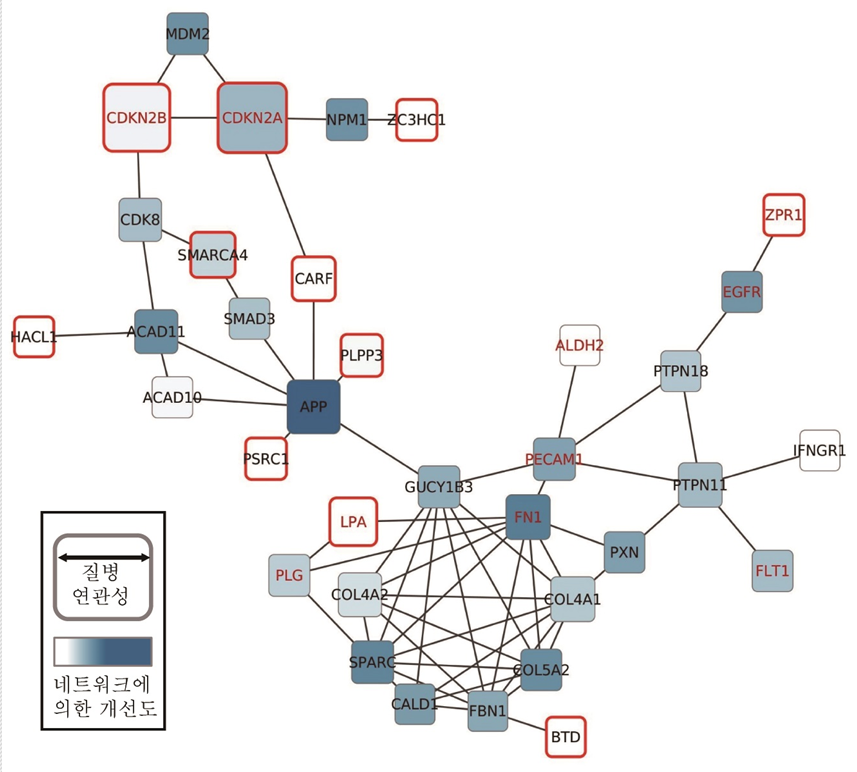
[그림2] 유전체코호크 빅데이터에서 도출된 심장동맥질환(Cardiac Artery Disease) 관련 유전자들의 네트워트 분석
연구팀은 코호트기반 GWAS에서 우선적으로 도출된 후보 질병유전자들이 유전자네트워크에서 서로 잘 연결되는 경향을 보인다는 기존의 관찰 결과를 근거로 삼았다. 나아가 GWAS 결과에서 통계적 유의성이 낮은 후보유전자들을 유의미한 질병유전자들과 네트워크상에서의 연결 정도를 이용하여 다시 발굴하는 방법을 개발했다. 또한 이를 개방형 분석 시스템인 GWAB(www.inetbio.org/gwab)으로 개발하여 국내외 모든 연구자들이 자신들의 유전체코호트 빅데이터를 인터넷을 통해 자유롭게 분석할 수 있도록 함으로써 코호트기반 질병유전체 연구에 큰 기여를 할 것으로 기대된다. 한편, 관련 논문은 해당 연구 분야의 권위 있는 국제 학술지 ‘뉴클레익 애시드 리서치(Nucleic Acids Research)’에 최근 게재되었다.
- vol. 전체
- vol. 635
- vol. 634
- vol. 633
- vol. 632
- vol. 631
- vol. 630
- vol. 629
- vol. 628
- vol. 627
- vol. 626
- vol. 625
- vol. 624
- vol. 623
- vol. 622
- vol. 621
- vol. 620
- vol. 619
- vol. 618
- vol. 617
- vol. 616
- vol. 615
- vol. 614
- vol. 613
- vol. 612
- vol. 611
- vol. 610
- vol. 609
- vol. 608
- vol. 607
- vol. 606
- vol. 605
- vol. 604
- vol. 603
- vol. 602
- vol. 601
- vol. 600
- vol. 599
- vol. 598
- vol. 597
- vol. 596
- vol. 595
- vol. 594
- vol. 593
- vol. 592
- vol. 591
- vol. 590
- vol. 589
- vol. 587
- vol. 586
- vol. 585
- vol. 584
- vol. 583
- vol. 582
- vol. 581
- vol. 580
- vol. 579
- vol. 578
- vol. 577
- vol. 576
- vol. 575
- vol. 574
- vol. 573
- vol. 572
- vol. 571
- vol. 570
- vol. 569
- vol. 568
- vol. 567
- vol. 566
- vol. 565
- vol. 564
- vol. 563
- vol. 562
- vol. 561
- vol. 560
- vol. 559
- vol. 558
- vol. 557
- vol. 556
- vol. 555
- vol. 554
- vol. 553
- vol. 552
- vol. 551
- vol. 550
- vol. 549
- vol. 548
- vol. 547
- vol. 546
- vol. 545
- vol. 544
- vol. 543
- vol. 542
- vol. 541
- vol. 540
- vol. 539
- vol. 538
- vol. 537
- vol. 536
- vol. 535
- vol. 534
- vol. 533
- vol. 532
- vol. 531
- vol. 530
- vol. 529
- vol. 528
- vol. 527
- vol. 526
- vol. 525
- vol. 524
- vol. 523
- vol. 522
- vol. 521
- vol. 520
- vol. 519
- vol. 518
- vol. 517
- vol. 516
- vol. 515
- vol. 514
- vol. 513
- vol. 512
- vol. 511
- vol. 510
- vol. 509
- vol. 508
- vol. 507
- vol. 506
- vol. 505
- vol. 504
- vol. 503
- vol. 502
- vol. 501
- vol. 500
- vol. 499
- vol. 498
- vol. 497
- vol. 496
- vol. 495
- vol. 494
- vol. 493
- vol. 492
- vol. 491
- vol. 490
- vol. 489
- vol. 488
- vol. 487
- vol. 486
- vol. 485
- vol. 484
- vol. 483
- vol. 482
- vol. 481
- vol. 480
- vol. 479
- vol. 478
- vol. 477
- vol. 476
- vol. 475
- vol. 474
- vol. 473
- vol. 472
- vol. 471
- vol. 470
- vol. 469
- vol. 468
- vol. 467
- vol. 466
- vol. 465
- vol. 464
- vol. 463
- vol. 462
- vol. 461
- vol. 460
- vol. 459
- vol. 458
- vol. 457
- vol. 456
- vol. 455
- vol. 454
- vol. 453
- vol. 452
- vol. 451
- vol. 450
- vol. 449
- vol. 448
- vol. 447
- vol. 446
- vol. 445
- vol. 444
- vol. 443
- vol. 442
- vol. 441
- vol. 440
- vol. 439
- vol. 438
- vol. 437
- vol. 436
- vol. 435
- vol. 434
- vol. 433
- vol. 432
- vol. 431
- vol. 430
- vol. 429
- vol. 428
- vol. 427
- vol. 426
- vol. 425
- vol. 424
- vol. 423
- vol. 422
- vol. 421
- vol. 420
- vol. 419
- vol. 418
- vol. 417
- vol. 416
- vol. 415
- vol. 414
- vol. 413
- vol. 412
- vol. 411
- vol. 410
- vol. 409
- vol. 408
- vol. 407
- vol. 406
- vol. 405
- vol. 404
- vol. 403
- vol. 402
- vol. 401
- vol. 400
- vol. 399
- vol. 398
- vol. 397
- vol. 396
- vol. 395
- vol. 394
- vol. 393
- vol. 392
- vol. 391
- vol. 390
- vol. 389
- vol. 388
- vol. 387
- vol. 386
- vol. 385
- vol. 384
- vol. 383
- vol. 382
- vol. 381
- vol. 380
- vol. 379
- vol. 378
- vol. 377
- vol. 376
- vol. 375
- vol. 374
- vol. 373
- vol. 372
- vol. 371
- vol. 370
- vol. 369
- vol. 368
- vol. 367
- vol. 366
- vol. 365
- vol. 364
- vol. 363
- vol. 362
- vol. 361
- vol. 360
- vol. 359
- vol. 358
- vol. 357
- vol. 356
- vol. 355
- vol. 354
- vol. 353
- vol. 352
- vol. 351
- vol. 350
- vol. 349
- vol. 348
- vol. 347
- vol. 346
- vol. 345
- vol. 344
- vol. 343
- vol. 342
- vol. 341
- vol. 340
- vol. 339
- vol. 338
- vol. 337
- vol. 336
- vol. 335
- vol. 334
- vol. 333
- vol. 332
- vol. 331
- vol. 330
- vol. 329
- vol. 328
- vol. 327
- vol. 326
- vol. 325
- vol. 324
- vol. 323
- vol. 322
- vol. 321
- vol. 320
- vol. 319
- vol. 318
- vol. 317
- vol. 316
- vol. 315
- vol. 314
- vol. 313
- vol. 312
- vol. 311
- vol. 310
- vol. 309
- vol. 308
- vol. 307
- vol. 306
- vol. 305
- vol. 304
- vol. 303
- vol. 302
- vol. 301
- vol. 300
- vol. 299
- vol. 298
- vol. 297
- vol. 296
- vol. 295
- vol. 294
- vol. 293
- vol. 292
- vol. 291
- vol. 290
- vol. 289
- vol. 288
- vol. 287
- vol. 286
- vol. 285
- vol. 284
- vol. 283
- vol. 282
- vol. 281
- vol. 280
- vol. 279
- vol. 278
- vol. 277
- vol. 276
- vol. 275
- vol. 274
- vol. 273
- vol. 272
- vol. 271
- vol. 270
- vol. 269
- vol. 268
- vol. 267
- vol. 266
- vol. 265
- vol. 264
- vol. 263
- vol. 262
- vol. 261
- vol. 260
- vol. 259
- vol. 258
- vol. 257
- vol. 256
- vol. 255
- vol. 254
- vol. 253
- vol. 252
- vol. 251
- vol. 250
- vol. 249
- vol. 248
- vol. 247
- vol. 246
- vol. 245
- vol. 244
- vol. 243
- vol. 242
- vol. 241
- vol. 240
- vol. 239
- vol. 238
- vol. 237
- vol. 236
- vol. 235
- vol. 234
- vol. 233
- vol. 232
- vol. 231
- vol. 230
- vol. 229
- vol. 228
- vol. 227
- vol. 226
- vol. 225
- vol. 224
- vol. 223
- vol. 222
- vol. 221
- vol. 220
- vol. 219
- vol. 218
- vol. 217
- vol. 216
- vol. 215
- vol. 214
- vol. 213
- vol. 212
- vol. 211
- vol. 210
- vol. 209
- vol. 208
- vol. 207
- vol. 206
- vol. 205
- vol. 204
- vol. 203
- vol. 202
- vol. 201
- vol. 189
- vol. 188
- vol. 187
- vol. 186
- vol. 185
- vol. 184
- vol. 183
- vol. 182
- vol. 181
- vol. 180
- vol. 179
- vol. 178
- vol. 177
- vol. 176
- vol. 175
- vol. 174
- vol. 173
- vol. 172
- vol. 171
- vol. 170
- vol. 169
- vol. 168
- vol. 167
- vol. 166
- vol. 165
- vol. 164
- vol. 163
- vol. 162
- vol. 161
- vol. 160
- vol. 159
- vol. 158
- vol. 157
- vol. 156
- vol. 155
- vol. 154
- vol. 153
- vol. 152
- vol. 151
- 전체 글 보기
- 연세 뉴스
- 연세통합 60주년
- 윤동주 탄생 100주년
- 섬김의 리더십
- 세계 in 연세
- 창업톡톡
- 칭찬편지공모전
- 화제의 인물
- 연구 프론티어
- 주니어 연구톡톡
- 하늘과바람과별과시
- 신촌캠퍼스 소식
- 의료원 소식
- 원주캠퍼스 소식
- 국제캠퍼스 소식
- 신간 안내
- 동정
- 공연 소식
- 지금 SNS에서는
연세소식 신청방법
아래 신청서를 작성 후 news@yonsei.ac.kr로 보내주세요신청서 다운로드